От биологии к биоинформатике: практическое руководство для новичков
Биоинформатика набирает обороты популярности по мере того, как биология осваивает так называемые «омики» — новое поколение данных. Термин «омики» (от англ. omics) обозначает комплекс современных направлений биологической науки, таких как геномика, транскриптомика, протеомика, метаболомика, названия которых имеют суффикс —омик. В результате все больше биологов обращаются к биоинформатике: одни — из любопытства, другие — в силу профессиональной необходимости, а третьи — из жизненной потребности. Однако биологов-новичков нередко страшит углубление в такие области, как программирование, статистика и анализ данных, тем более что в учебном плане по биологии этим дисциплинам уделено довольно скромное место.
Если вы, будучи биологом, хотите заняться биоинформатикой, но не знаете, с чего начать, не паникуйте! Сегодня благодаря огромному количеству доступных ресурсов и инструментов сделать это проще, чем когда-либо. К тому же я дам вам рекомендации, которые помогли мне самому преодолеть путь от биологии к полноценной биоинформатике. Начнем с основ.
Освойте соответствующую среду (программирования)
Возможно, вы знаете (если нет, готовьтесь к плохой новости), что большинство биоинформационных инструментов разрабатываются для использования в операционной системе (ОС) на базе Linux, поэтому вам необходимо освоить эту среду и ее команды (язык программирования Bash). При отсутствии доступа к серверу (внешнему компьютеру, предоставляющему возможность проводить анализы), на котором наверняка установлен Linux, вам придется создать собственную среду.
Если вы пользователь Mac, вам повезло! И macOS, и Linux относятся к одному семейству ОС (Unix), что позволит вам использовать терминал на macOS для запуска большинства необходимых команд и программ Bash. Открывайте терминал и начинайте практиковаться! Если же вы являетесь пользователем Windows, придется немного больше потрудиться, чтобы настроить свою среду. Хорошая новость заключается в том, что у вас будет несколько вариантов.
Отличный способ для пользователей Windows полностью погрузиться в среду Linux — установить дружественный дистрибутив Ubuntu. Если же вы слишком привыкли к Windows (понимаю, как трудно расстаться с этой ОС, особенно если вы истинный фанат компьютерных игр), не обязательно отказываться от дорогого компаньона. Можете загрузить полноценную Ubuntu вместе с Windows (путем двойной загрузки), установить Ubuntu на виртуальную машину (проще говоря, компьютер внутри компьютера) или воспользоваться подсистемой Windows для Linux, чтобы получить простую установку Ubuntu с терминалом Linux для начала работы. Многочисленные руководства, которые легко найти в интернете, помогут вам справиться с любым из этих трех вариантов.
Теперь перейдем к самому программированию. Вы можете создавать программы даже в приложении Notepad (забудьте, что я это сказал) или в редакторах вроде Vim или Nano, которые хорошо известны в сфере Linux. Но они (очень) сложны в освоении, поэтому лучше использовать их только для быстрого редактирования. Почему бы не сделать жизнь максимально простой и комфортной?
Я бы рекомендовал использовать такой удобный редактор, как Visual Studio Code — самый популярный и предпочитаемый мной лично. Ключевая особенность этого инструмента — обширная экосистема плагинов, которые позволяют настроить его по своему вкусу и облегчить жизнь программиста. Подсветка синтаксиса? — Есть. Завершение кода? — Присутствует. Подключение к удаленному серверу (если он у вас есть) и редактирование файлов там? — Без проблем. Сопровождающие текст пиксельные питомцы? — Исключительны. Редактор может быть настолько удобен, насколько вы захотите.
Visual Studio Code поддерживает большинство языков программирования, включая наиболее применяемые в биоинформатике Bash и Python. Что же касается языка R, который вам наверняка пригодится и который также поддерживается в Visual Studio Code, возможно, понадобится использовать среду разработки RStudio с встроенными в нее специальными функциями для R, наиболее удобными для анализа данных.
Выбирайте наименьшее зло, извлекая из него наибольшую пользу.
Следуйте лучшим практикам при создании кода
Это общий совет по программированию вдвойне актуален для биоинформатики: программирование — не просто создание кода. Хотя языки программирования допускают большую гибкость в написании кода, существуют определенные техники, позволяющие сделать его более читаемым и удобным для сопровождения.
Каждый язык имеет свой набор руководств по стилю, и следование им — хорошая привычка, которую следует выработать с самого начала. Например, в Python основным руководством по стилю является PEP 8. Подобные руководства создаются не просто так: следуя им, вы обеспечиваете согласованность в написании кода, что облегчает его применение другими пользователями. Недаром говорится:
«Код чаще читают, чем пишут».
Учитывая это, старайтесь писать чистый и последовательный код, который говорит сам за себя.
Не менее важно документирование кода. Речь идет о строках, которые допускаются в языках программирования, но не выполняются компьютером, поскольку предназначены для читателя. Например, в Python, Bash и R строки, начинающиеся с #, являются комментариями. Комментарии должны сопровождать любой сложный или трудный для понимания код, чтобы объяснить его назначение.
Адекватное документирование кода, которое иногда игнорируется, поскольку требует времени, — не напрасный труд: читая, используя или изменяя код, сопровождаемый корректными комментариями, другие программисты смогут отточить свои навыки. Более того, и что очень важно, если вам понадобится вернуться к своему коду спустя долгое время (две недели — иногда большой срок, в зависимости от вашей памяти), вы поблагодарите себя за документирование (см. изображение ниже).

Наконец, контроль версий и резервное копирование — обязательное условие для любого проекта, который вам дорог. Git позволяет отслеживать изменения, которые вы вносите в код, и возвращаться к предыдущим версиям, если что-то пойдет не так (спойлер: так и будет).
Git создает репозиторий («repository») в папке вашего проекта, где хранятся изменения, которые вы вносите в свои файлы. Вы будете добавлять («add») и коммитить («commit») эти изменения в репозитории с сообщениями, объясняющими внесенные в код поправки. Затем сможете перенести («push») эти изменения в удаленный репозиторий для резервного копирования или сделать их общедоступными (например, на GitHub). Являясь отличным инструментом для личных проектов, Git особенно полезен для работы в команде. Хотя это довольно сложный инструмент, базовое понимание его основных команд — таких как add, commit и push — облегчит ваше продвижение в программировании. С ним вы забудете о форс-мажорных ситуациях типа:
«Упс, я удалил всю свою работу без резервной копии!»
Имейте под рукой (виртуальный) блокнот
Биологи в лабораторных условиях ведут специальные журналы. Почему бы и вам не завести свой блокнот (notebook)?
Запись полученных биоинформационных данных сделает вашу работу (и обучение) намного эффективнее. Какие анализы я запустил в этот день? Какие программы использовал? С какими параметрами? Какие результаты получил? Возможно, в краткосрочной перспективе на эти вопросы будет легко ответить. Но со временем накопление анализов, результатов, многочисленных проектов, предупреждений, ошибок, критических замечаний перегрузит вас информацией.
Обычного блокнота будет достаточно, но, поскольку вы работаете в компьютерной среде, приложение для ведения заметок на рабочем столе может быть гораздо удобнее. В качестве примера можно привести Obsidian — текстовый редактор заметок, который использует синтаксис Markdown — простой и легкий в освоении язык разметки для форматирования текста. Заметки в виде обычного текста отлично подходят для копирования и вставки команд, фрагментов кода и результатов. Кроме того, приложения для ведения заметок со встроенными функциями поиска делают доступ к этой информации быстрым и удобным. Как и в случае с Visual Studio Code, многие из них предлагают плагины для дальнейшей настройки виртуального блокнота и даже подключения к соответствующим приложениям, таким как Zotero.
Блокнот для биоинформатики позволит вам:
- Собрать наиболее часто используемые команды (в виде шпаргалки).
- Составить собственный глоссарий биоинформационных терминов по обработке или анализу данных.
- Записывать способы предупреждения ошибок, с которыми вы сталкиваетесь, для дальнейшего использования.
- Фиксировать результаты анализов с вашими умозаключениями и интерпретациями.
Возможности безграничны, так что подумайте над тем, как использовать виртуальный блокнот в своих целях.
Изучите способы конвейеризации
Возможно, в биоинформатике нет термина «конвейеризация» («pipelining»). Биоинформационный анализ чаще всего предполагают последовательное выполнение набора команд с очень четко определенными входными (input) и выходными (output) данными. Эти шаги записываются (обычно на языке Bash) в так называемые конвейеры (pipelines), позволяющие автоматизировать анализ с помощью одного выполнения программы.
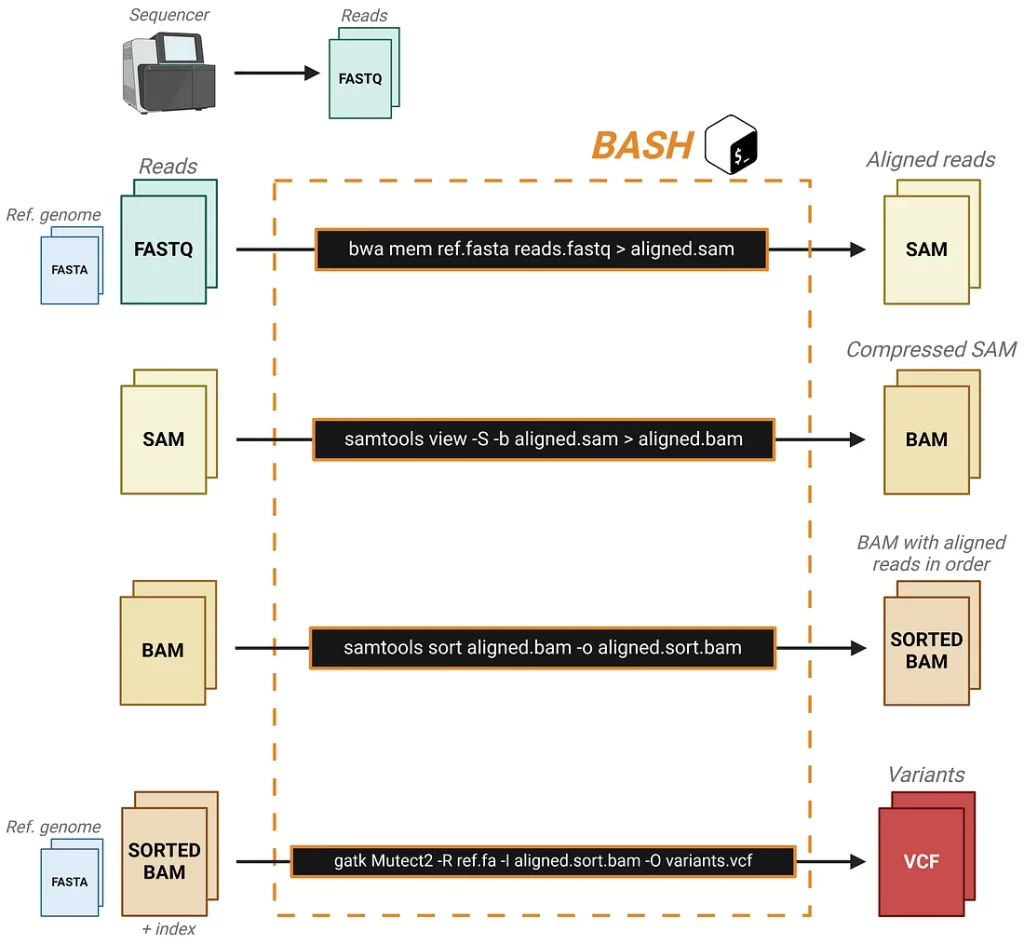
Возможность автоматизировать процессы — один из самых мощных инструментов биоинформатиков, и вы должны использовать его в полной мере. При необходимости выполнить один и тот же набор команд несколько раз записывайте их для автоматической реализации в конвейере. Записав команды в файл, вы сможете выполнить его, чтобы легко повторить тот же самый анализ, скорректировать команды при необходимости или осуществить отладку, если все идет не так, как планировалось.
Менеджеры рабочих процессов, такие как Nextflow или Snakemake, — еще один шаг на пути к созданию конвейера, который позволит вам спать по ночам (по крайней мере, на первых этапах). Это самостоятельные языки программирования, помогающие более эффективно организовывать выполнение конвейеров, например, с точки зрения оптимизации распределения вычислительных ресурсов и времени вычислений. Менеджеры рабочих процессов также способствуют беспрепятственному выполнению конвейеров в различных вычислительных средах (локальных/серверных, кластерных, облачных) и повышают их воспроизводимость. Пока что помните об их существовании и преимуществах, которые они дают, чтобы в будущем изучить их.
Но упомянув Nextflow, не могу не сказать о связанной с ним работе сообщества nf-core, которая представляет для вас особый интерес. nf-core предлагает большой набор тщательно подобранных и готовых к использованию конвейеров, написанных на Nextflow, обеспечивая конвейеризированное выполнение большинства распространенных омиксных анализов (определение вариантов, РНК-секвенирование, полногеномное секвенирование с предварительной обработкой бисульфитом и многих других). Их можно легко запустить, выполнив несложные требования по установке Nextflow и настройке параметров конвейера (часто минимальных), чтобы провести анализ в соответствии с конкретным данным. При использовании конвейеров у вас не возникнет проблем, потому что они отлично документированы (вы уже знаете, как это важно), а сообщество открыто для обратной связи через Slack-канал.
Конвейеры nf-core хорошо воспроизводятся, оптимизированы для производительности, предоставляют замечательные отчеты и поддерживаются сообществом экспертов, которые следят за тем, чтобы конвейеры соответствовали современным стандартам. Они станут вашим подспорьем при необходимости развертывания конвейера для высокостандартного омиксного анализа. Можете считать nf-core полезным обучающим ресурсом, позволяющим получить представление о ключевых шагах и лучших практиках для каждого типа анализа.
Управляя зависимостями, стремитесь к воспроизводимости
Версии программ и пакетов, используемых в биоинформационном анализе, имеют значение. Программы меняют свое поведение от одной версии к другой, а это означает, что результаты, получаемые в том или ином анализе, привязаны к конкретной среде, в которой вы работали. Хорошо бы иметь для каждого проекта автономную среду, в которой программы и их версии четко определены.
Менеджеры пакетов, такие как Conda, невероятно полезны для управления средами. Conda позволяет создавать по требованию «виртуальные среды» — по сути, изолированные коллекции программ, которые можно загружать и выгружать по мере необходимости. Отличаясь простотой использования, Conda с помощью одной команды устанавливает необходимое программное обеспечение — от пакетов Python и R до полноценных программ, — а также все ПО, от которого они зависят.
После завершения проекта вы сможете поделиться рецептом создания такой среды с другими программистами, чтобы они могли воспроизвести вашу работу или сохранить ваш рецепт для использования в будущем.
Еще один шаг — контейнеры, которыми можно управлять, в частности, с помощью Docker или Singularity. Каждый контейнер — не только автономная (кто бы мог подумать!) среда, которая может выполняться, упаковывая установленные в него программы, но и своего рода «мини-ОС», что еще больше повышает воспроизводимость. Если вы поделитесь этим контейнером с коллегами (например, в Docker Hub), они смогут выполнить те же команды, что и вы, с теми же результатами, независимо от базовой среды разработки.
Как и в случае с менеджерами рабочих процессов, не призываю вас немедленно приступить к освоению контейнеров. Хочу только заметить, что оба эти инструмента станут бесценными по мере вашего карьерного продвижения. Оба они, что очень важно, направлены на повышение воспроизводимости работы биоинформатика (как и любого другого ученого). Делая небольшие шаги в сторону открытости (например, выкладывая свой код на GitHub) и воспроизводимости (указывая среду, использованную в ваших анализах), вы встанете на верный путь осуществления своей миссии как биоинформатика.
Не забывайте о биологии в [био]информатике
Работу биоинформатика иногда понимают превратно. По общему мнению, он должен обладать солидным опытом в области компьютерных наук и иметь дело почти исключительно с компьютерами, получая травмы рук не от интенсивного пипетирования, а от интенсивного щелканья мышью. Даже само слово «биоинформатика» на 79 % состоит из информатики и лишь на 21 % — из биологии. Такое отношение к профессии может сбить вас с толку, заставив думать, что биоинформатика — только информатика, но это не так. Напротив, реальность такова, что биологическая часть, в конце концов, является сердцем биоинформатики. Просто биоинформатик изучает иначе биологию.
Не позволяйте себя ввести в заблуждение — работая с массивами данных, довольно легко потеряться в море цифр и букв. Особенно это касается биоинформатиков, которые не работают в условиях лабораторий, поскольку существует разрыв между созданием данных и самими данными. Вам может казаться, что данные волшебным образом появляются в вашем компьтере: раз — и вот они уже на экране! Но биологические данные — гораздо больше, чем просто цифры и буквы: они являются показателями биологического процесса. И, как таковые, должны обрабатываться, анализироваться и интерпретироваться с учетом биологического контекста.
При работе над каждым конкретном проектом вы должны задать себе следующие вопросы:
- Каков биологический источник моих данных?
- Какой экспериментальный протокол использовался для их получения?
- Как этот протокол влияет на выбор способа обработки данных? Какие инструменты и статистические методы лучше всего подходят для моих данных?
- Какой анализ следует провести, чтобы ответить на поставленный мной биологический вопрос?
- Составляют ли мои результаты целостную картину с учетом природы образца и имеющихся у нас биологических знаний?
Не проводите анализ бездумно: биологический контекст данных должен быть центральным пунктом вашего анализа.
Идите в ногу со временем (чего бы это ни стоило)
Биоинформатика зависит от двух постоянно развивающихся областей (биологии и информатики), поэтому изменения в ней происходят так быстро, как только возможно. Биологические знания постоянно расширяются: публикуются результаты новых исследований, возникают новые вопросы, разрабатываются новые методы для ответа на них, и вместе с ними кардинально меняется наше понимание биологических процессов. Биоинформационные инструменты и технологии неотъемлемо связаны с этим циклом изменений, одновременно подвергаясь его влиянию и внося в него свой вклад. Да и сами они находятся в состоянии перманентного изменения: постоянно разрабатываются новые инструменты и технологии, обновляются прежние (с разной степенью частоты). Но это еще не все.
Основа биоинформатики — создание кода — также эволюционирует, и, возможно, сейчас, как никогда раньше, в связи с появлением инструментов, основанных на искусственном интеллекте. Создание кода ни в коем случае не является таким, каким было 10 лет назад, и, несомненно, не будет таким же через 10 лет. Генеративные модели сегодня позволяют генерировать и завершать код с помощью простых описаний, что не только снижает барьер для входа в программирование, но и дает возможность тем, кто уже владеет им, делать больше за меньшее время. Время, которое необходимо, чтобы заниматься биологией.
Из всего этого следует, что биоинформатикам необходимо идти в ногу со многими изменениями во многих сферах. И, возможно, лучший совет, который я могу вам дать, заключается в следующих словах: постарайтесь сделать все возможное, чтобы справиться с этой сложнейшей задачей. Читайте статьи, следите за ведущими фигурами в этой области, посещайте конференции, участвуйте в семинарах и, самое главное, будьте открыты всему новому. Не отвергайте происходящие вокруг вас изменения только потому, что они не привычны для вас: примите их. Помните завет легендарного Брюс Ли:
«Будь как вода, мой друг».
Будьте всегда гибкими, готовыми адаптироваться к новым способам программирования и методам биоинформатики, которые становятся неотъемлемой частью этой области.
Читайте также:
- 10 бесплатных Mac-приложений, о которых вы точно не знали
- Raspberry Pi + Pushover + Puppeteer = автоматизация повседневных задач
- 7 недооцененных GitHub-проектов, которые могут быть полезны
Читайте нас в Telegram, VK и Дзен
Перевод статьи Daniel G. Camblor: Take-off from Biology to Bioinformatics: A Practical Guide for Newcomers